• Luca Scapoli1
• Ambra Girardi1
• Dorina Lauritano2
• Annalisa Palmieri1
1Dipartimento di Medicina Specialistica, Diagnostica e Sperimentale, Università di Bologna, Italia
2Dipartimento di Medicina e Chirurgia Interdisciplinari, Università di Milano-Bicocca, Milano, Italia
Riassunto
La malattia parodontale distrugge i tessuti che sostengono gli elementi dentari. Il danno tissutale è causato da una risposta infiammatoria prolungata ad un cambiamento microbiologico nella composizione dei biofilm sottogengivale.
Le tre specie batteriche che costituiscono il complesso gruppo rosso, Porphyromonas gingivalis, Tannerella forsizia e Treponema denticola, sono considerate i principali patogeni coinvolti nella parodontite.
Nel presente studio è stato progettato una saggio real-time PCR per rilevare e quantificare le specie del “Red Complex” al fine di indagare 307 campioni di tasche parodontali da 127 pazienti affetti da parodontite e da 180 controlli. I risultati hanno dimostrato una significativa prevalenza di specie del “red complex e una maggiore quantità di P. gingivalis e T. denticola nelle tasche parodontali dei pazienti affetti da parodontite.
Summary
Innovative technologies in clinical practice: a modern diagnostic-therapeutic approach to periodontal disease.
Periodontitis is a disease mainly caused by a chronic infection of tissues that support teeth. Several factors, such as diabetes, smoking and oral care, as well as genetic susceptibility can influence both the risk to develop periodontitis and its progression. The aim of the investigation was to test whether alleles of candidate genes were associate to periodontitis. A case control study was performed with a cohort of 182 patients with chronic periodontitis and 230 healthy controls from the Italian population. A total of six single nucleotide polymorphisms from five candidate genes, i.e. IL1A, IL1B, IL6, IL10 and VDR, were investigated. Evidence of association were obtained for rs1800795 mapping in IL6 (P value = 0.01), as well as for the rs1800872 mapping in IL10 (P = 0.04). The rarer variant allele lowered the risk to develop periodontitis at IL6 OR = 0.69 (95% C.I. 0.51-0.93), while increased the risk at IL10 OR = 1.38 (1.01-1.86). The present investigation indicated that polymorphisms of IL6 and IL10 constitute risk factors for chronic periodontitis, while there was no evidence implicating a specific IL-1A or IL-1B genotype.
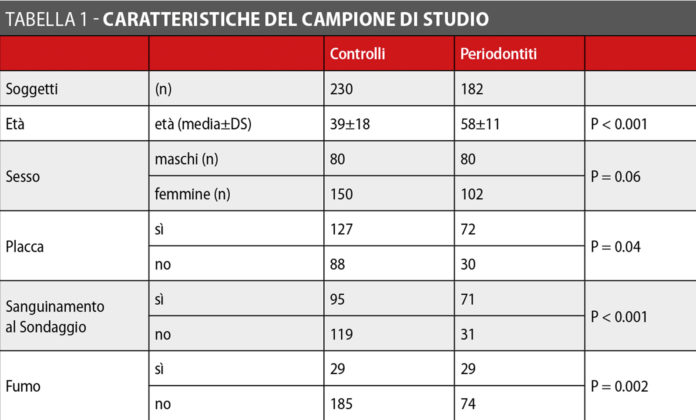
La malattia parodontale è una patologia multifattoriale, in cui sia fattori ambientali che genetici giocano un ruolo preciso e controverso nel determinarne l’insorgenza. La flora batterica orale riveste sicuramente un ruolo importante nella progressione di tale patologia. Ulteriori fattori di rischio, ampiamente studiati, sono il fumo e il diabete. Tuttavia, una serie di fattori genetici dell’ospite possono condizionare la suscettibilità individuale all’insorgenza della malattia, determinarne le differenti manifestazioni cliniche e la velocità di progressione1.
A differenza delle malattie genetiche mendeliane, che sono rare e causate da una singola o poche mutazioni, le malattie multifattoriali, quali la parodontite, sono frequenti e correlate a numerosi fattori ambientali e genetici. I fattori genetici non sono vere e proprie mutazioni, ma polimorfismi genetici, detti anche fattori di suscettibilità. Ognuno di questi non è necessario, né sufficiente, a determinare la malattia, tuttavia risulta in grado di modificarne il rischio di insorgenza.
Questi polimorfismi sono variazioni a livello del codice genetico che possono avere diversi effetti, per esempio cambiare i livelli di espressione genica, causare lievi modifiche funzionali delle molecole codificate, rendendo gli individui più suscettibili all’insorgenza di una determinata malattia o al manifestarsi di quadri clinici più gravi della malattia stessa2.
Negli ultimi anni le indagini sui fattori di suscettibilità allo sviluppo della malattia parodontale si sono indirizzate principalmente allo studio di geni che codificano fattori coinvolti nella modulazione della risposta immunitaria, recettori della superficie cellulare, chemochine, enzimi e proteine correlate al riconoscimento dell’antigene. Le citochine, come IL-1A, IL-1B, IL-10 e IL-6, sono fattori chiave che mediano il processo infiammatorio nella malattia parodontale. Esse hanno un ruolo nell’attivazione, nella proliferazione e nella differenziazione delle cellule B, le principali cellule implicate nelle manifestazioni severe di periodontite3.
Tali variazioni genetiche possono perciò favorire la progressione della malattia4 causandone il classico andamento, caratterizzato da cicli ripetuti di infiammazione tissutale, seguiti da remissioni spontanee (definito andamento a “pousses”)5.
Nella malattia parodontale i batteri patogeni accumulati nel solco subgengivale sono i fattori ambientali che influenzano la risposta infiammatoria dei tessuti parodontali6. Tuttavia, le citochine sono considerate anch’esse indirettamente responsabili della distruzione del tessuto connettivo e del riassorbimento osseo7.
Dal momento che il riassorbimento osseo alveolare è un fattore chiave nella malattia parodontale, il recettore della vitamina D (VDR) è stato considerato come un fattore di suscettibilità nella progressione della malattia. Dati in letteratura sostengono l’esistenza di un’associazione tra comuni polimorfismi a carico di geni candidati e la malattia parodontale8,9.
È interessante rilevare che la maggior parte degli studi genetici sulla periodontite ha impiegato coorti di piccole dimensioni. Il limitato potere statistico di studi condotti con un basso numero di campioni conduce a un’imprecisa valutazione del livello di rischio genetico e al pericolo di ottenere risultati falsi positivi e falsi negativi.
Ciò spiega anche il fatto che spesso tali studi sono giunti a opposte conclusioni. Un’accurata selezione di un elevato numero di campioni è dunque un requisito necessario per rilevare correttamente un’eventuale associazione tra polimorfismi genetici e insorgenza di parodontite. Inoltre, il numero e il tipo di geni che influenzano l’andamento della malattia parodontale potrebbero essere variabili in relazione all’etnia della popolazione studiata e al grado di severità della malattia.
Nel presente lavoro abbiamo analizzato sei specifici polimorfismi a carico dei geni codificanti per IL-1A, IL-1B, IL-6, IL10 e VDR, al fine di verificare se essi agiscano come fattori di suscettibilità della malattia parodontale cronica nella popolazione italiana.
Materiali e metodi
Pazienti
Per questo studio sono stati reclutati 412 soggetti; di questi, 182 erano affetti da malattia parodontale cronica, mentre il gruppo di controllo era costituito da 230 adulti sani o affetti da lieve gengivite.
La Tabella 1 riassume le principali caratteristiche e i dati clinici rilevati nell’ambito dei gruppi.
Il prelievo biologico è stato eseguito dalla tasca parodontale di un singolo sito mediante l’introduzione di un cono di carta utilizzando il LABtest (per gentile concessione della LAB® srl, Codigoro, Ferrara).
Il DNA è stato estratto e purificato utilizzando un protocollo standard che prevede l’incubazione con proteinasi K, seguite da una purificazione mediante spin-column.
Genotipizzazione
Sono stati investigati i seguenti sei polimorfismi: per IL1A il NM_000575.3:c.-949C>T (rs1800587); per IL1B il NM_000576.2:c.315C>T (rs1143634); per IL6 il XR_108749.1:n.50-321G>C (rs1800795);
per IL10 il NG_012088.1:g.3943A>G (rs1800896) e NG_012088.1:g.4433A>C (rs1800872); per VDR il NM_000376.2:c.1056T>C (rs731236).
I saggi sono stati selezionati utilizzando l’Applied Biosystems SNPbrowser Software (Life Technologies, Milano).
I genotipi per tutti i polimorfismi sono stati ottenuti con utilizzo dello strumento ABI PRISM 7500 Sequence Detection System
e il metodo TaqMan, secondo i protocolli forniti dall’azienda (Life Technologies, Milano).
I genotipi sono stati raccolti dai ricercatori in un sistema cieco (i biologi non avevano a disposizione le informazioni cliniche dei pazienti da cui provenivano i campioni analizzati).
Analisi statistica
La distribuzione dei genotipi nei pazienti e nei controlli è stata testata per le deviazioni dall’equilibrio Hardy-Weinberg utilizzando il test del χ2. L’associazione genetica è stata valutata con l’impiego di test genotipici e allelici con un approccio “likelihood ratio” utilizzando il software Unphased v3.1.5 in un sistema operativo Windows Vista10.
Sono stati calcolati gli odds ratio per valutare gli effetti dell’allele raro nei portatori, sia per gli eterozigoti che per gli omozigoti. L’associazione degli aplotipi è stata valutata per il cluster IL1 (IL1A e IL1B) e IL10 perché erano disponibili due polimorfismi tra loro vicini. In particolare, è stato eseguito un test di associazione globale, così come un test di associazione specifico per ciascun aplotipo.
Risultati
Il protocollo per l’analisi genetica del DNA genomico del paziente, ottenuto e purificato dal fluido crevicolare, si è rivelato molto efficace. Infatti, il tasso di successo della genotipizzazione dei 6 differenti polimorfismi era compreso tra il 96% e il 99%.
Solo 20 campioni di DNA su 412 hanno prodotto un numero di genotipi pari o inferiore a tre su sei.
Le frequenze genotipiche sono risultate essere in accordo con l’equilibrio di Hardy-Weinberg sia nel campione intero sia limitatamente a ciascun gruppo (i casi e i controlli).
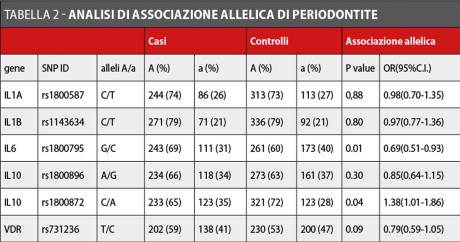
Il confronto tra le frequenze alleliche osservate nei casi e nei controlli è riportato nella Tabella 2.
Si è osservata una differenza significativa, tra casi e controlli, per i polimorfismi rs1800795 dell’IL6 (P = 0,01), e rs1800872 dell’IL10 (P = 0.04). Valori di associazione marginali sono stati ottenuti invece per lo SNP rs731236 di VDR (P = 0.09).
In effetti, le persone portatrici dell’allele raro per le varianti a carico di IL6 e VDR sono risultate essere meno suscettibili alla parodontite, con valori di OR = 0,69 (IC 95% 0,51-0,93) e OR = 0,79 (IC 95% 0,59-1,05), rispettivamente. L’allele raro per IL10 ha mostrato invece un aumento di rischio OR = 1,38 (IC 95% 1,01-1,86).
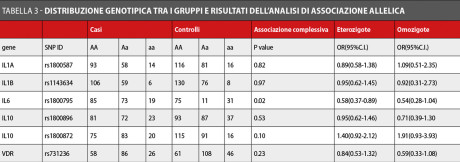
Il test di associazione genotipica ha fornito risultati equivalenti (Tabella 3).
Il polimorfismo rs1800795 di IL6 ha mostrato una significativa associazione genotipica globale (valore di P = 0.02) e un minor rischio per i portatori dell’allele raro sia in eterozigosi che in omozigosi, con un OR = 0,58 (IC 95% 0,37-0,89) e OR = 0,54 (IC 95% 0,28-1,04), rispettivamente.
Valori, questi, vicini ai livelli nominali di significatività.
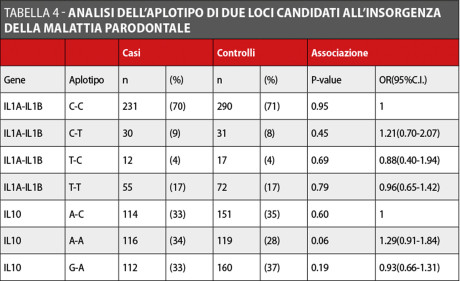
Sono stati evidenziati alti livelli di linkage disequilibrium fra gli SNPs del cluster di geni dell’IL1 (D’.= 0,75; r2 = 0,44) e tra gli SNPs del gene IL10 (D’ = 0.93; r2 = 0,22).
Questo ci ha permesso di combinare i dati di due marcatori adiacenti per eseguire l’analisi dell’associazione aplotipica.
L’analisi statistica complessiva, per testare se gli aplotipi fossero complessivamente associati alla parodontite, ha evidenziato la mancanza di associazione sia con il cluster genico dell’IL1 (P = 0,87), sia con il locus dell’IL10 (P = 0.16).
I valori specifici di associazione e gli odds ratio stimati per ciascun aplotipo sono riportati in Tabella 4.
L’aplotipo rs1800896(A)-rs1800872(A) ha mostrato un livello di associazione con la parodontite borderline OR = 1,29 (IC 95% 0,91-1,84), p-value = 0,06.
Discussione
Molti studi hanno valutato l’associazione tra varianti genetiche e malattia parodontale, tuttavia non vi è ancora un accordo tra gli studiosi per quanto riguarda le correlazioni tra basi genetiche e manifestazioni cliniche di questa patologia.
Infatti, i numerosi studi hanno spesso riportato risultati contrastanti a causa, principalmente, della diversa etnia delle popolazioni studiate, ma anche delle piccole dimensioni dei campioni oggetto di studio. Infatti tali studi – con meno di 100 pazienti e 100 controlli – non sono in grado di fornire una potenza statistica in grado di rilevare un moderato effetto genetico, cioè un odds ratio di 1,5 o inferiore.
Un’altra fonte di variabilità potrebbe essere correlata alle diverse finalità delle indagini, essendo a volte mirate a focalizzare l’associazione tra polimorfismi genetici e fenotipi specifici, come la parodontite aggressiva o la parodontite cronica, oppure l’associazione con la differente risposta ai trattamenti clinici. Qui abbiamo riportato i risultati di uno studio genetico effettuato con 182 pazienti affetti da parodontite cronica e 230 controlli, tutti di origine italiana. Il fluido crevicolare raccolto con una sonda di carta è stato usato come fonte di DNA genomico.
L’efficienza del test genetico su questo DNA è risultata alta, in effetti ha fornito circa il 98% dei genotipi. I risultati delle analisi di associazione genetica dei loci differenti sono discussi di seguito per ciascun locus.
IL1A e IL1B
IL1A e IL1B gene e sette altri geni della famiglia dell’interleuchina 1 formano un cluster genico delle citochine sul cromosoma 2. I polimorfismi a carico di IL1A e IL1B sono stati tra i primi e i maggiormente indagati per l’associazione con la parodontite. Nel presente lavoro non è stata trovata evidenza di associazione tra rs1800587 (noto anche come IL1A-889C>T), rs1143634 (anche conosciuto come IL1B 3954 C>T) e la malattia. Degni di nota sono i valori di odds ratio rilevati, poiché erano prossimi a 1 sia nell’analisi di associazione allelica che nell’analisi di associazione genotipica; i risultati suggeriscono che non vi è alcuna tendenza all’associazione. Risultati simili sono stati ottenuti quando entrambi i polimorfismi sono stati analizzati insieme nell’analisi degli aplotipi (Tabella 4).
In una recente review che considerava indagini condotte con almeno 100 individui sia nei casi che nei controlli, gli Autori hanno concluso che i polimorfismi del cluster genico IL1 non possono essere considerati come fattori di rischio o di suscettibilià di parodontite aggressiva o cronica9. I nostri risultati, ottenuti su un campione di popolazione italiana, giungono alla stessa conclusione. D’altra parte, una meta-analisi che combinava i dati di 13 studi effettuati su pazienti caucasici affetti da malattia parodontale cronica ha rilevato correlazioni significative per le due varianti geniche individuali (IL1A OR = 1.48 e IL1B OR = 1.54). Considerando che la dimensione del campione del nostro studio è in grado di individuare tale livello di associazione (calcolo non riportato) si può ipotizzare che questo contrasto sia riconducibile a eterogeneità genetica tra le diverse popolazioni caucasiche, dato che i polimorfismi dell’IL1 non sono risultati associati alla malattia parodontale nella popolazione italiana. Altri studi condotti con campioni di popolazione Italiana riportano assenza di associazione con la parodontite aggressiva11.
IL6
I polimorfismi mappanti in IL6 sono ripetutamente segnalati come fattori di rischio sia per la parodontite cronica che aggressiva in diverse popolazioni; altri studi invece mostrano un’associazione negativa9. Conseguentemente gli Autori della rassegna affermano che nessuna conclusione certa si può trarre riguardo questo locus.
Nel nostro studio il livello più forte di associazione è stato trovato in questo locus, infatti la variante allelica rs1800795-C (polimorfismo noto anche come IL6-174G>C) era significativamente meno rappresentato nei pazienti affetti da malattia parodontale. Questo sta a indicare che i portatori dell’allele rs1800795-C presentavano rischio minore, pari circa alla metà, di sviluppare malattia parodontale come precedentemente osservato da altri ricercatori9.
IL10
È stato ipotizzato che IL10 possa rallentare la distruzione del tessuto parodontale attraverso l’induzione di inibitori tissutali delle metalloproteinasi e dell’osteoprotegerina, noto inibitore di osteoclastogenesi12.
Nel nostro studio sono stati indagati due polimorfismi della regione a monte del gene IL10 che ne regola i livelli di espressione. È stato osservato che la variante allelica rs1800872-A aumentava il rischio di sviluppare parodontite con un OR osservato di 1,38 (IC 95% 1,01-1,86).
È interessante notare che Reichert et al. hanno dimostrato che questo allele è funzionale nello sviluppo della malattia cronica, essendo associato a una ridotta espressione di IL1013.
VDR
Il recettore della vitamina D (VDR) regola una varietà di processi biologici, compresi il metabolismo osseo e la risposta immunitaria alle infezioni microbiche14. Visto che il riassorbimento osseo alveolare e l’accumulo di batteri sono le caratteristiche principali della malattia parodontale, è possibile che il recettore della vitamina D (VDR) e i suoi polimorfismi genetici giochino un ruolo nella suscettibilità all’insorgenza di malattia parodontale.
Nel presente studio è stato osservato un livello di associazione tra VDR e parodontite al limite della significatività statistica.
Un’indagine supplementare con indipendenti ulteriori campioni o polimorfismi diversi potrà fornire ulteriori informazioni per chiarire il ruolo di VDR nella parodontite.
Conclusioni
In questo studio è stata dimostrata un’associazione statisticamente significativa tra le varianti alleliche comuni, IL6 rs1800795-G e IL10 rs1800872-A, e la malattia parodontale.
Questi dati supportano l’uso di questi polimorfismi in un test basato sul DNA per migliorare la diagnostica della malattia parodontale.
Ulteriori sviluppi del nostro studio potrebbero consentire un’applicazione nella pratica clinica dell’Odontoiatra.
Conoscere il profilo genetico del paziente, o in altre parole la sua predisposizione alla malattia, potrebbe risultare di grande utilità nella diagnostica della malattia parodontale e nella definizione di un piano terapeutico personalizzato. Inoltre, potrebbe dare indicazioni prognostiche sull’esito della malattia.
Corrispondenza
Annalisa Palmieri
Dipartimento di Medicina Specialistica, Diagnostica e Sperimentale
Università di Bologna
Via Belmeloro, 8 – Bologna
annalisa.palmieri@unife.it
Ringraziamenti
Questo lavoro è stato sostenuto da LAB srl, Codigoro (Fe), Italia.
Bibliografia
1. Heitz-Mayfield LJ. Disease progression: identification of high-risk groups and individuals for periodontitis. J Clin Periodontol 2005;32 Suppl 6:196-209.
2. Craandijk J, van Krugten MV, Verweij CL, van der Velden U, Loos BG. Tumor necrosis factor-alpha gene polymorphisms in relation to periodontitis. J Clin Periodontol 2002;29:28-34.
3. Yamazaki K, Nakajima T, Gemmell E, Polak B, Seymour GJ, Hara K. IL-4- and IL-6-producing cells in human periodontal disease tissue. J Oral Pathol Med 1994;23:347-53.
4. Scarel-Caminaga RM, Trevilatto PC, Souza AP, Brito RB, Jr., Line SR. Investigation of IL4 gene polymorphism in individuals with different levels of chronic periodontitis in a Brazilian population. J Clin Periodontol 2003;30:341-5.
5. Duff GW. Molecular genetics of cytokines: Cytokines in chronic inflammatory disease. In: Thompson, editor. The cytokine handbook. London: Academic Press., 1998:21-33.
6. Wilson M, Reddi K, Henderson B. Cytokine-inducing components of periodontopathogenic bacteria. J Periodontal Res 1996;31:393-407.
7. Okada H, Murakami S. Cytokine expression in periodontal health and disease. Crit Rev Oral Biol Med 1998;9:248-66.
8. Karimbux NY, Saraiya VM, Elangovan S, Allareddy V, Kinnunen T, Kornman KS, et al. Interleukin-1 gene polymorphisms and chronic periodontitis in adult caucasians: a systematic review and meta-analysis. J Periodontol 2012 Nov;83(11):1407-19.
9. Laine ML, Loos BG, Crielaard W. Gene polymorphisms in chronic periodontitis. Int J Dent 2010:324719.
10. Dudbridge F, Gusnanto A. Estimation of significance thresholds for genomewide association scans. Genet Epidemiol 2008;32:227-34.
11. Scapoli C, Borzani I, Guarnelli ME, Mamolini E, Annunziata M, Guida L, et al. IL-1 gene cluster is not linked to aggressive periodontitis. J Dent Res 2010;89(5):457-61.
12. Claudino M, Trombone AP, Cardoso CR, Ferreira SB Jr., Martins W Jr., Assis GF, et al. The broad effects of the functional IL-10 promoter-592 polymorphism: modulation of IL-10, TIMP-3, and OPG expression and their association with periodontal disease outcome. J Leukoc Biol 2008;84:1565-73.
13. Reichert S, Machulla HK, Klapproth J, Zimmermann U, Reichert Y, Glaser CH, et al. The interleukin-10 promoter haplotype ATA is a putative risk factor for aggressive periodontitis. J Periodontal Res 2008;43:40-7.
14. Amano Y, Cho Y, Matsunawa M, Komiyama K, Makishima M. Increased nuclear expression and transactivation of vitamin D receptor by the cardiotonic steroid bufalin in human myeloid leukemia cells. J Steroid Biochem Mol Biol 2009;114:144-51.